Estudo preliminar de um modelo em Excel para auxiliar na identificação de Enterobacterales para usuários de provas bioquímicas manuais
Preliminary study of an Excel model to assist in the identification of Enterobacterales for users of manual biochemical tests
Alessandra Fagundes Bonetti1
1 Universidade Federal do Rio Grande do Sul – UFRGS, Especialização em Microbiologia Clínica. Porto Alegre, RS – Brasil.
Recebido em 09/10/2023
Aprovado em 03/12/2023
DOI: 10.21877/2448-3877.202300151
INTRODUÇÃO
Os bacilos Gram-negativos pertencentes à ordem Enterobacterales são os isolados mais frequentemente encontrados em espécimes clínicos analisados no setor de microbiologia de um laboratório de análises clínicas. Para sua identificação são utilizados métodos convencionais (manuais), automatizados e, mais atualmente, a espectrometria de massa por ionização/dessorção a laser auxiliada por matriz (MALDI-TOF MS). Os métodos automatizados oferecem muitas vantagens como, por exemplo, a reprodutibilidade e a disponibilidade de fornecer resultados dentro de 24 horas. Além disso, esses métodos apresentam um sistema de interfaceamento que facilita ao médico obter resultados parciais ou integrais do exame de seu paciente.(1) As desvantagens, porém, são o custo elevado e a dependência de um sistema fechado, que exige manutenção e renovação de seus softwares.(2)
A tecnologia proteômica, com a utilização da metodologia de MALDI-TOF MS, deu início a uma nova era, de modo que novos gêneros foram validados e muitas novas espécies foram descritas.(3) No entanto, os custos da implementação da tecnologia de espectrofotometria de massas ainda estão além das possibilidades financeiras de muitos laboratórios.
Os laboratórios de pequeno e médio portes que não contam com métodos automatizados fazem a identificação de Enterobacterales através de testes bioquímicos manuais, nos quais características metabólicas específicas são detectadas nos microrganismos. Após a leitura dos testes, os resultados são interpretados através de consulta em tabelas. Tais tabelas são fornecidas pelas principais literaturas de referência em microbiologia e possuem a caracterização bioquímica dos principais microrganismos, o que permite a identificação da bactéria a partir da coincidência do resultado com o perfil fornecido para espécie/gênero. Com este método, é possível realizar a identificação dos principais gêneros bacterianos de interesse clínico(4) a um custo mais acessível para os laboratórios com menos recursos, embora tal rotina seja problemática em razão da falta de praticidade, principalmente para os profissionais com menor experiência, pois é necessário o manuseio físico de tabelas extensas, demandando demasiado tempo.
É relevante observar que os métodos fenotípicos de identificação microbiana, como os testes bioquímicos manuais, ainda são os métodos de primeira escolha para a realização do diagnóstico microbiológico na maioria dos laboratórios dos países em desenvolvimento.(5) Dados do PNCQ – Programa Nacional de Controle de Qualidade da Sociedade Brasileira de Análises Clínicas, referentes ao ano de 2022, apontam para a mesma realidade: aproximadamente 87% das identificações encaminhadas para o Programa utilizaram métodos manuais, ou seja, foram 23.037 cepas identificadas pelo método manual versus 3.405 pelo método automatizado.(6)
OBJETIVO
Elaborar um modelo informatizado em planilha Excel, como alternativa dinâmica de identificação bacteriana para uso em laboratórios de pequeno e médio portes que utilizam os testes bioquímicos Citrato, Ureia, Ornitina, Lisina, TSI, SIM, para identificação de Enterobacterales de forma manual.
MATERIAIS E MÉTODOS
Inicialmente, foi desenvolvida uma planilha no modelo Excel 2016 em que os testes bioquímicos (Citrato, Ureia, Ornitina, Motilidade, H2S, Lisina, Lactose, Indol, Gás) foram organizados em colunas. Para cada teste foram atribuídas possibilidades de resultado positivo ou negativo usando a função SE do Excel como condicionante (SE(teste_lógico;[valor_se_verdadeiro]; [valor_se falso]).(7)
Um banco de dados com 512 possibilidades de perfis para as diferentes famílias e gêneros bacterianos de maior probabilidade de isolamento em laboratórios de menor porte foi inserido conforme literatura especializada.(4,8) A planilha foi desenvolvida também com a capacidade de calcular a probabilidade conforme frequência de ocorrência nos testes de identificação dos microrganismos. Para este cálculo foi necessário, além da tabela de resultado dos perfis para cada família e gênero bacteriano, fornecer ao modelo em Excel, a tabela com os perfis de positividade para cada teste bioquímico.(4,8)
A identificação de uma bactéria desconhecida foi baseada, então, no cálculo da probabilidade entre o perfil desconhecido e o perfil de cada espécie de microrganismo armazenada no banco de dados da planilha Excel 2016.(8) Os cálculos seguiram o modelo exemplificado por Koneman et al, 2001,(8) em que somente a probabilidade de reação positiva da prova listada na base de dados é utilizada para cálculo. A probabilidade da reação negativa é 1 menos a probabilidade da reação positiva. A frequência de ocorrência é calculada por multiplicação conjunta de todas as frequências de ocorrência das reações. Por fim, para obtenção da porcentagem de identificação, cada frequência é dividida pela soma de todas as frequências, e o resultado é multiplicado por 100.
As identificações que atingiram uma probabilidade de 90% ou mais foram consideradas excelentes identificações, enquanto aquelas que se aproximaram de 90% foram consideradas satisfatórias, porém com a recomendação de que fossem submetidas a melhor identificação por meios de provas adicionais ou liberação da identificação somente em nível de gênero. Por fim, foram desconsideradas aquelas com probabilidade muito baixa.(8)
Após concluída a etapa do desenvolvimento da planilha, esta foi validada comparando os resultados encontrados com aqueles obtidos pelo método referência de interpretação. Neste método, os testes são lidos e comparados visualmente com os resultados constantes em uma tabela de reações bioquímicas conhecidas para cada família e/ou gênero.
As amostras utilizadas para validação da planilha foram de isolados clínicos (urinas) e isolados provenientes do Controle Externo de Qualidade (PNCQ). Os resultados obtidos com a rotina clássica e com o modelo Excel foram também comparados com resultados obtidos por MALDI-TOF MS.
Os testes bioquímicos manuais e as placas com meio de cultivo primário utilizados para cultura e identificação de Enterobacterales foram aqueles utilizados na rotina do Laboratório Núcleo Análises Clínicas/Gravataí e adquiridos da marca Laborclin®. São eles: Citrato de Simmons, Triple Sugar Iron (TSI), Lisina Descarboxilase (LIA), Ornitina Descarboxilase (MIO), Ágar Ureia, SIM, Ágar Mueller Hinton, assim como placa de Ágar MacConkey.
As respostas bioquímicas proporcionadas pelo conjunto de meios identificatórios foram:(9)
- Citrato de Simmons: utilização do citrato de sódio como única fonte de carbono;
- Meio SIM: produção de H2S, produção de indol (após adição de reativo de Kovacs), motilidade;
- Meio de MIO: descarboxilacão da ornitina, produção de indol (após adição de reativo de Kovacs) e motilidade;
- Meio de LIA: descarboxilação da lisina, desaminação da lisina, produção de H2S;
- Meio de TSI: fermentação da glicose, fermentação da lactose, produção de H2S e gás (quanto a esta prova, em especial, deve ser registrado o fato de que o TSI detecta lactose e sacarose, de forma que poderá ocorrer positividade na superfície do meio de organismos fermentadores da sacarose e não fermentadores da lactose, sendo que se faz necessária a confirmação da fermentação ou não da lactose através de meios seletivos como o Mac Conkey ou, ainda, a utilização da Prova de Kliger Iron Agar, em que o diferencial para o Triple Sugar Iron Agar – TSI é a ausência da sacarose).
- Urease (Meio de Christensen): produção de urease (nesta prova, deve ser destacada a importância de se usar o Meio de Christensen, em vez da Ureia em caldo, visto que esta última só detecta microrganismos fortes produtores de urease, como o gênero Proteus, por exemplo, e não consegue detectar fracos produtores, como, por exemplo, o gênero Klebsiella sp.).
A semeadura, temperatura e tempo de incubação, anteriores à leitura dos resultados, seguiram a orientação presente na bula do fornecedor e também presente no Procedimento Operacional Padrão do Laboratório (POP).
Cabe ressaltar que boas práticas em laboratório de microbiologia são importantes e foram seguidas, como por exemplo: não foram usadas colônias isoladas em um período superior a 24 horas, pois, a partir deste período, o metabolismo bacteriano pode ficar comprometido e a leitura de alguns parâmetros pode consequentemente ficar prejudicada ou até mesmo não ocorrer; assim como não foram usadas colônias muito recentes (inferior ao período de 18 horas), pois não se encontram com o metabolismo bem definido e algumas provas podem ter seus resultados alterados; a leitura das provas foi realizada dentro de um período de 18 a 24 horas; aguardou-se que os meios de cultura de identificação bacteriana estivessem em temperatura ambiente para então se realizar os testes; assim como não foi usado sobrecarga de inóculo.(9,10)
As identificações bacterianas obtidas através da metodologia de MALDI-TOF MS foram realizadas de acordo com instruções do fabricante, no Laboratório de Pesquisa em Resistência Bacteriana (LABRESIS-HCPA). O software utilizado para a identificação foi o MBT Compass 4.1 (Bruker).
As cepas foram acondicionadas em eppendorf com caldo TSB (Laborclin®) com 16% de glicerol e armazenadas em freezer no período de março de 2023 até junho do mesmo ano.
RESULTADOS
As funções implementadas na planilha conseguiram associar, após a introdução dos resultados encontrados no bioquimismo bacteriano, a identificação automatizada de microrganismo nos isolados testados (Figura 1).
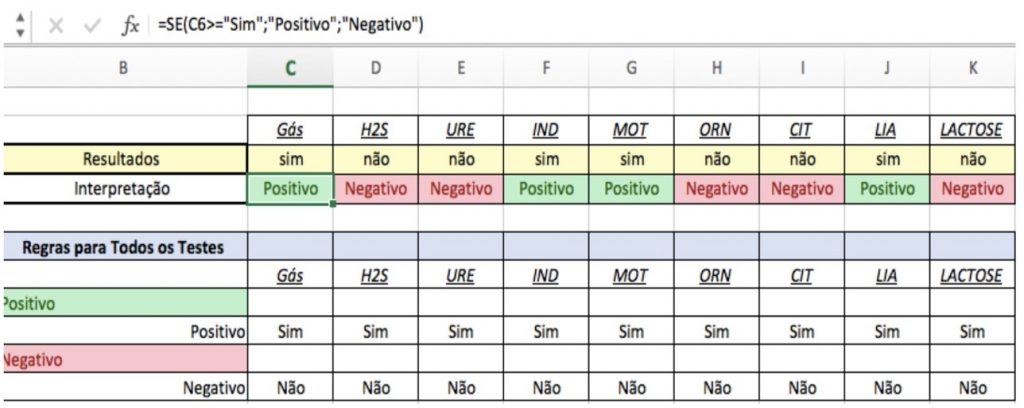
Figura 1
Tabela Excel – Exemplo de atribuição de resultados positivos ou negativos aos testes bioquímicos e função SE.
Fonte: Elaborado pela autora (2023).
Com a implementação do banco de dados com as tabelas com todos os perfis de resultados para os testes bioquímicos da ordem Enterobacterales, além da tabela com o perfil de positividade frente aos testes e os cálculos de probabilidade, a planilha conseguiu identificar o microrganismo e fazê-lo em ordem de probabilidade (Figuras 2,3,4).
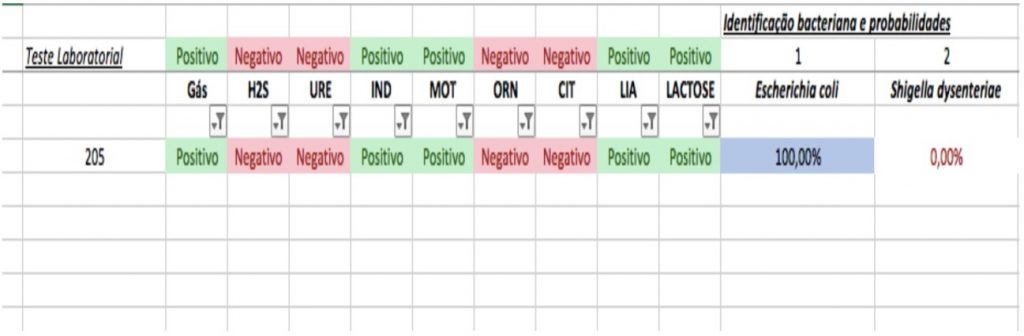
Figura 2
Tabela Excel- Exemplo de identificação bacteriana e probabilidade.
Fonte: Elaborado pela autora (2023).
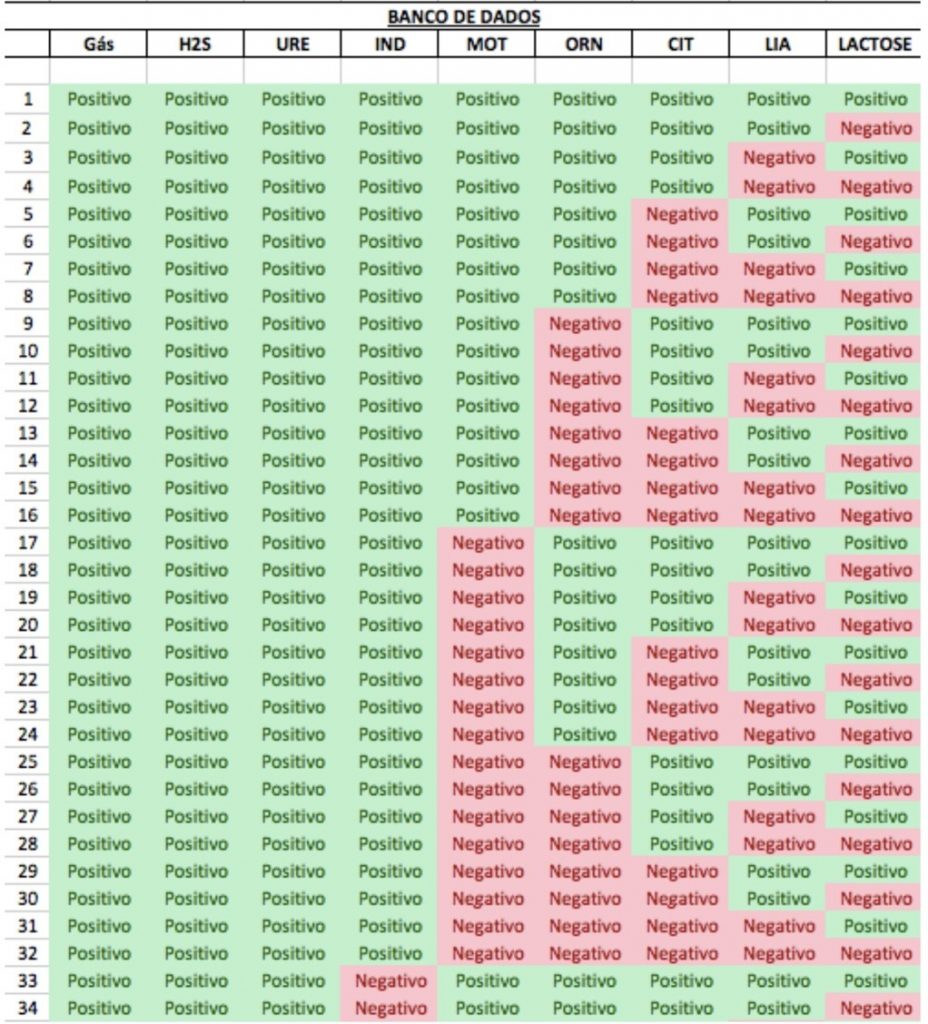
Figura 3
Banco de Dados – Exemplo de tabela de perfil de positividade frente aos testes bioquímicos dos gêneros e espécies bacterianas.
Fonte: Elaborado pela autora (2023).
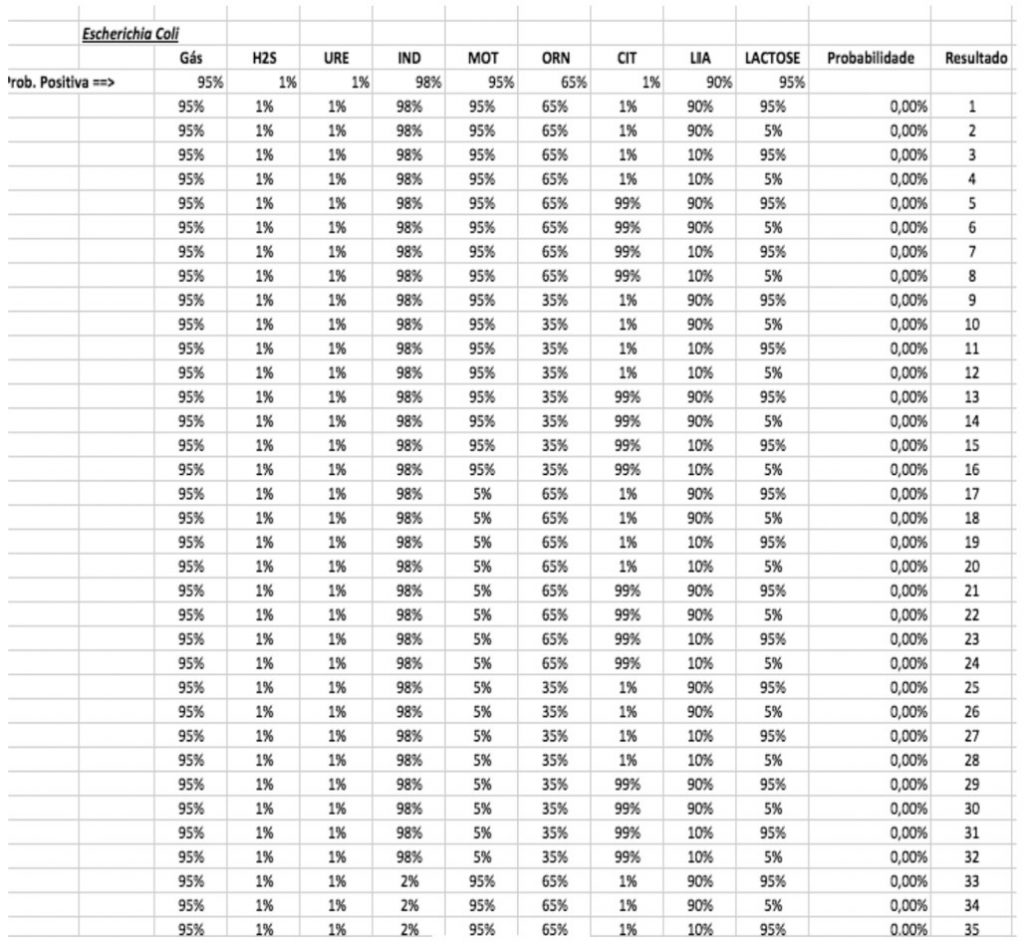
Figura 4
Banco de dados – Exemplo de tabela de frequência de positividade perante os testes bioquímicos de cada gênero e espécie bacteriana.
Fonte: Elaborado pela autora (2023).
A validação da planilha Excel foi feita mediante comparação com os resultados obtidos através do método referência de interpretação, bem como, igualmente, com os resultados obtidos por MALDI-TOF MS, utilizando isolados de amostras clínicas de urina e cepas provenientes do Controle Externo de Qualidade (PNCQ).
Foram 32 amostras que apresentaram identidade com alto índice de probabilidade (superior a 90%). Duas amostras (21 e 22) de Escherichia coli não apresentaram resultado esperado para sua espécie, ou seja, não foram capazes de utilizar o carboidrato lactose presente no meio, como característico, sendo desta forma somente possível a identificação com o uso da tabela Excel ou pelo MALDI-TOF MS.
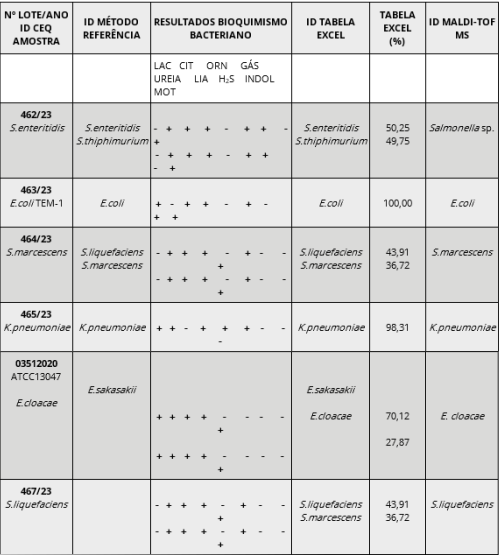
De outra parte, quando testadas as 6 bactérias provenientes do Programa Nacional de Controle de Qualidade (PNCQ), 2 destes microrganismos (Escherichia coli Tem-1 e Klebsiella pneumoniae) foram identificados conforme o método referência, sendo que tais identificações atingiram um bom índice de probabilidade (superior a 90%). As outras 3 bactérias (Salmonella enteritidis; Serratia marcescens e Serratia liquefaciens) foram identificadas com mais de uma possibilidade de identificação bacteriana, o que também se verificou tanto no método clássico como no método de leitora em planilha Excel, porém a identificação mediante leitura na tabela Excel possibilitou concluir pela necessidade de execução de mais provas bioquímicas para determinação da bactéria (dada a baixa probabilidade) ou liberação do resultado apenas pelo gênero bacteriano.
A amostra fornecida pelo Controle Externo de Qualidade (PNCQ) lote 03512020/ATCC 13047 de Enterobacter cloacae não apresentou resultado esperado para sua espécie, ou seja, não foi capaz de hidrolisar a ureia presente no meio através da enzima urease, como característico, sendo, assim, somente possível a identificação pelo MALDI-TOF MS. Com o uso da tabela Excel, o Enterobacter cloacae foi identificado com um percentual de 27,87%, enquanto o Enterobacter sakasakii obteve 70,12%.
Registra-se, como premissa, o fato de que a tabela, quando preenchida com os resultados esperados pela literatura, guardou correspondência com o microrganismo correto, o que evidencia que as eventuais limitações decorrem de fatores que lhe são externos.
Por fim, quando comparados os resultados obtidos na identificação pelo método clássico com os resultados da planilha Excel e com os resultados obtidos no MALDI-TOF MS, verificou-se que somente 4 amostras (11, 12, 25 e 26) de Klebsiella variicola foram identificadas equivocadamente como Klebsiella oxytoca, tanto pelo método referência como pela leitura com a planilha Excel. O microrganismo em questão faz parte do Complexo Klebsiella pneumoniae, o qual teve sua taxonomia expandida recentemente e que somente pode ser identificado com o auxílio dos métodos moleculares, genômicos, proteônicos ou pela atualização dos protocolos desenvolvidos para aumentar a precisão na identificação de novas espécies de tal Complexo.
Nas Tabelas 1 e 2, seguem os resultados encontrados para as amostras isoladas e identificadas de urinas da rotina do laboratório e de cepas do Controle Externo de Qualidade (PNCQ).
DISCUSSÃO
Segundo o Programa Nacional de Controle de Qualidade da Sociedade Brasileira de Análises Clínicas,(6) a maioria dos laboratórios que participam do controle externo utiliza métodos manuais de identificação para Enterobacterales. O objetivo deste trabalho foi proporcionar, aos microbiologistas que utilizam tais métodos, uma alternativa mais dinâmica para realizar a identificação bacteriana a partir dos resultados obtidos nos testes, mediante simples lançamento dos mesmos em planilha Excel que fará a identificação, inclusive com os percentuais correspondentes, reduzindo o tempo necessário para realização da tarefa.
O objetivo do trabalho foi alcançado, embora seja necessário registrar que o método proposto não afasta a influência de fatores externos e independentes da planilha Excel, que ocorreriam igualmente sem a sua utilização, bem como, ainda, que os resultados encontrados foram de um número relativamente pequeno de isolados, quando a família Enterobacterales possui inúmeros gêneros e espécies, algumas humanas e muitas ambientais, sendo a tabela proposta somente de utilização em isolados clínicos.
Dentre tais fatores externos, podemos mencionar os meios identificatórios para bacilos Gram-negativos pertencentes à família das Enterobacterales, que possuem limitações inerentes ao método, como, por exemplo, o fato de que a positividade de 90% de algumas bactérias a determinado teste exige uma incubação em até 48 horas, que nem sempre é cumprida. É o caso do meio citrato, por exemplo, no qual os produtos metabólicos decorrentes da reação provocam alcalinização do meio de cultura, que é detectado pelo indicador de pH incluído na formulação em até 48 horas.(9)
Na execução deste trabalho, em caso de reação negativa nas primeiras 24 horas, principalmente em identificações indeterminadas, os tubos foram mantidos em incubação por 24 horas adicionais, tendo sido considerado positivo quando o meio citrato mudou de cor para azul (originalmente verde) ou foi observado crescimento na linha de semeadura, demonstrando que a bactéria foi capaz de utilizar o citrato como única fonte de carbono.(9)
A dificuldade de observação da resposta frente à motilidade bacteriana é outro complicador que pode ser melhorado com o entendimento sobre o movimento bacteriano, que é demonstrado através da turvação dos meios semissólidos SIM ou MIO a partir da localização do inóculo (Figura 5).

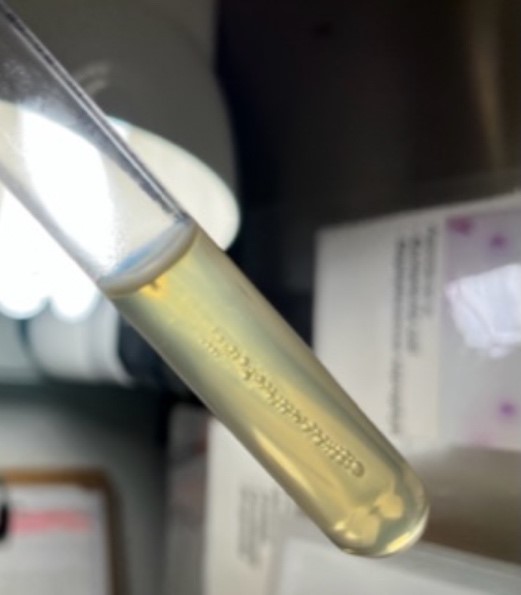
Figura 5
Motilidade bacteriana negativa e positiva no meio de SIM.
Fonte: Elaborado pela autora (2023).
Tabela 1
Resultados comparativos das identificações em isolados urinários feitas por metodologia referência, planilha Excel (objeto do trabalho) e MALDI-TOF MS.
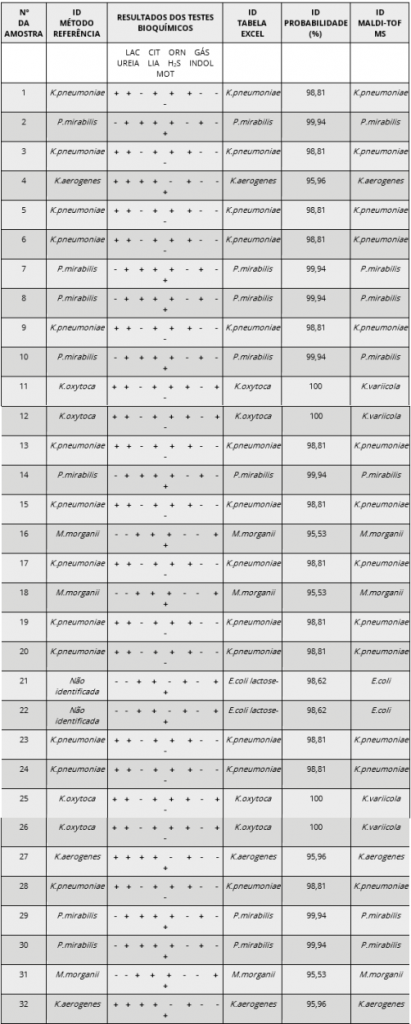
Tabela 2
Resultados comparativos das identificações em cepas CEQ-PNCQ feitas por metodologia referência, planilha Excel (objeto do trabalho) e MALDI-TOF MS.
Igualmente, o meio de identificação urease, que é um importante diferenciador entre a família dos Proteus e dos demais gêneros de Enterobacterales, também é um fator complicador, pois algumas Enterobacterales podem apresentar lenta atividade de urease, ou seja, são positivos tardios.(9)
É relevante salientar, acerca da validação realizada, que o Manual de Procedimentos Básicos em Microbiologia Clínica para o Controle de Infecção Hospitalar da Agência Nacional de Vigilância Sanitária do Ministério da Saúde (2002) registra que qualquer sistema de testes existentes no comércio, com leitura manual ou automatizada, apresenta limitações no número de provas e na discriminação dos diferentes gêneros e espécies de enterobactérias, de modo que a maioria dos esquemas trabalha com um máximo de 80% de acerto. É importante destacar que mesmo os sistemas semiautomatizados e automatizados não oferecem 100% de acerto para a caracterização das espécies de enterobactérias, mas representam o principal comportamento descrito na literatura.(10)
O mesmo Manual também registra que para as espécies dos gêneros Citrobacter, Enterobacter, Klebsiella e Serratia os testes mais utilizados apresentam baixo poder de discriminação, sendo a identificação feita pelo maior percentual de probabilidade.(10)
CONCLUSÃO
As funções implementadas na planilha conseguiram associar, após a introdução dos resultados encontrados no bioquimismo bacteriano, a identificação automatizada de microrganismo nos isolados testados.
Com a implementação do banco de dados com as tabelas com todos os perfis de resultados para os testes bioquímicos da ordem Enterobacterales, além da tabela com o perfil de positividade frente aos testes e os cálculos de probabilidade, a planilha conseguiu identificar o microrganismo e fazê-lo em ordem de probabilidade.
A validação levada a termo na execução do presente trabalho permite a conclusão de que a planilha em Excel desenvolvida apresenta índices de conformidade com o método clássico, sobressaindo-se pela maior dinâmica e facilidade de acesso aos resultados, com inegável ganho em termos de otimização das rotinas de trabalho.
Quando comparada com o método MALDI-TOF MS, a identificação pela tabela Excel apresentou discriminação inferior. Porém, a superioridade do método proteômico em termos de agilidade e acurácia, como alternativa para os laboratórios que possuem maior capacidade orçamentária, nunca foi alvo de questionamento no presente trabalho.
AGRADECIMENTOS
Ao Laboratório Núcleo Análises Clínicas, por disponibilizar os resultados de identificações de amostras de cepas bacterianas que foram de grande utilidade para a elaboração deste trabalho.
À biomédica responsável pelo Controle de Qualidade no Laboratório Núcleo, Fernanda Carpeggiani, pelas informações e estatísticas obtidas junto ao Programa Nacional de Controle de Qualidade- PNCQ.
Ao Laboratório de Pesquisa em Resistência Bacteriana (LABRESIS-HCPA) pela oportunidade de acesso às facilidades e recursos que muito enriqueceram este estudo.
REFERÊNCIAS
- Donay JL, Mathieu D, Fernandes P, Prégermain C, Bruel P, Wargnier A et al (2004). Evaluation of the automated phoenix system for potential routine use in the clinical microbiology laboratory. J Clin Microbiol 42(4):1542-6. https://doi.org/10.1128/JCM.42.4.1542-1546.2004
- Leo S, Cherkaoui A, Renzi G, Schrenzel J (2020). Mini Review: clinical routine Microbiology in the era of automation and digital health. Front Cell Infect Microbiol 10:582028. https://doi.org/10.3389/fcimb.2020.582028
- Greub G (2010). MALDI-TOF mass spectrometry: the quantum leap. Clin Microbiol Infect 16(11):1603. https://doi.org/10.1111/j.1469-0691.2010.03380.x
- Oplustil CP, Zoccoli CM, Tobouti NR, Scheffer MC (2020). Procedimentos básicos em Microbiologia Clínica. 4. ed. Sarvier, São Paulo.
- García Blancas Pablo, Mendoza Medellín Aurelio (2014). Pruebas bioquímicas tradicionales y de alta resolución para identificación manual de enterobacterias. Acta Bioquím. Clín. Latinoam 48(2):249-254. http://www.scielo.org.ar/scielo.php?script=sci_arttext&pid=S0325-29572014000200011. Acesso em: 10 Mar 2023.
- PNCQ – Programa Nacional de Controle de Qualidade (2023). https://pncq.org.br. Acesso em: 17 Mar 2023.
- Tostes PR (2016). Fórmulas, funções e matrizes no Excel 2016. 1. ed. Alta Books, Rio de Janeiro.
- Koneman EW, Allen D. Stephen, Janda M. William, Schreckenberger C. Paul, Winn Washington C Jr. (2001). Diagnóstico Microbiológico. 5. ed. Medsi, Rio de Janeiro.
- Antunes GS (1995). Manual de Diagnóstico Bacteriológico. 2. ed. Editora da UFRGS, Porto Alegre.
- Anvisa – Agência Nacional de Vigilância Sanitária (2020). Detecção e Identificação de Bactérias de Importância Médica. https://bvsms.saude.gov.br/bvs/publicacoes/manual_microbiologia_mod5.pdf. Acesso em: 23 Jun 2023.
Correspondência
Alessandra Fagundes Bonetti
E-mail: [email protected]
