Disseminação da resistência aos antimicrobianos no contexto de saúde única: uma breve revisão
Dissemination of resistance to antimicrobials in the unique health context: a brief review
Líllian Oliveira Pereira da Silva1, Letícia Brasil Estevam2, Joseli Maria da Rocha Nogueira3
1 Escola Nacional de Saúde Pública Sérgio Arouca, Doutorado em Saúde Pública e Meio Ambiente. Rio de Janeiro, RJ, Brasil.
2 Fundação Técnico-Educacional Souza Marques, Especialização em Análises Clínicas. Rio de Janeiro, RJ, Brasil.
3 Escola Nacional de Saúde Pública Sérgio Arouca, Departamento de Ciências Biológicas. Rio de Janeiro, RJ, Brasil.
Recebido em 03/10/2023
Aprovado em 23/01/2024
DOI: 10.21877/2448-3877.202400149
INTRODUÇÃO
Infecções por bactérias resistentes são uma das maiores preocupações de saúde pública mundial, haja vista o aumento dos custos dos pacientes, uma vez que os fármacos comercializados atualmente passam a ser considerados ineficazes.(1,2) Ainda que a antibioticoterapia tenha sido revolucionária, a pressão seletiva causada pela utilização incorreta desses fármacos acaba favorecendo o surgimento de mutações e, consequentemente, o avanço da resistência aos antimicrobianos (RAM).(3,4)
De acordo com o último relatório da Organização Mundial de Saúde (OMS), o Brasil está entre os 20 países que mais consomem antibióticos em todo o mundo, com uma média de 22 doses por cada mil habitantes, em que ao menos 50% dos antimicrobianos prescritos são dispensados e/ou utilizados de maneira incorreta.(5-7)
Por sua vez, a RAM causa cerca de 700 mil mortes anuais, cuja previsão era de mais de 10 milhões de mortes até 2050.(8) Contudo, ressalta-se que estes dados foram publicados antes da pandemia da Covid-19, a qual elevou o consumo de antimicrobianos no país.(4) É a partir dessa premissa que há uma grande preocupação com o agravamento da resistência bacteriana em todo o mundo e, por consequência, uma futura pandemia causada por microrganismos pan-resistentes.
Ainda que haja um estímulo crescente ao estudo e desenvolvimento de novos antimicrobianos, trata-se de um processo lento, necessitando de diversas etapas para se obter algum resultado conclusivo e de grande valia para a comunidade.(9) De acordo com a OMS, os antimicrobianos que estão sendo desenvolvidos ainda não são capazes de contornar a problemática relacionada às famílias bacterianas mais perigosas do mundo, que podem ser identificadas no Quadro 1.
Não obstante, a RAM pode impactar diretamente na saúde humana, animal e ambiental, conforme o conceito de Saúde única, uma vez que sua disseminação ocorre a partir de diferentes fontes e reservatórios, como a agricultura, principal área consumidora de antimicrobianos, e a pecuária, onde estes fármacos são utilizados não só para a prevenção e/ou tratamento de infecções, mas também para a promoção do crescimento dos animais.(11,12)
A partir disso, o presente trabalho tem como objetivo discorrer sobre as vias de disseminação dos genes de resistência aos antimicrobianos (ARG, do inglês antimicrobial resistant gene) e das bactérias resistentes aos antimicrobianos (ARB, do inglês antimicrobial resistant bacteria), explorando as possibilidades de uma nova pandemia causada por bactérias resistentes aos antimicrobianos, uma vez que a propagação destes microrganismos é facilitada pela globalização.
METODOLOGIA
A metodologia se baseou em uma revisão da literatura, por meio da busca de artigos e trabalhos acadêmicos que abordassem a relação entre a resistência aos antimicrobianos e as diferentes vias de disseminação dos genes de resistência. O levantamento foi realizado no PubMed e no Google Acadêmico, utilizando a seguinte estratégia de busca:
(Resistência Bacteriana) OR (Bacterial resistance) AND (Zoonose) OR (Zoonosis) AND (Disseminação) OR (Dissemination) AND (Saúde única) OR (One Health) AND (Peixes) OR (Fishes) OR (Aquacultura) OR (Aquaculture) OR (Piscicultura) OR (Pisciculture) AND (Animais domésticos) OR (Domestic animals) AND (Agricultura) OR (Agriculture) AND (Resistoma) OR (Resistome) AND (Efluentes) OR (Effluents) AND (Alimentos) OR (Foods)
Para abreviar o processo de busca e seleção dos artigos, foram estipulados atalhos metodológicos, conforme o Quadro 2.
Para a seleção dos artigos, optou-se pela presença das palavras-chave, leitura dos títulos, seguida da leitura dos resumos e leitura na íntegra, onde os trabalhos que apresentavam relação com a pergunta de pesquisa do presente estudo; 35 artigos foram selecionados para compor os resultados desta revisão.
Quadro 1
Agentes patogênicos prioritários para o desenvolvimento de novos antibióticos.
| Prioridade 01: Crítica | Acinetobacter baumannii, resistente a carbapenêmicos |
| Pseudomonas aeruginosa, resistentes a carbapenêmicos | |
| Enterobacterales, resistente a carbapenêmicos, produtoras de ESBL (beta-lactamases de espectro estendido) | |
| Prioridade 02: Alta | Enterococcus faecium, resistente à vancomicina |
| Staphylococcus aureus, resistente à meticilina, com sensibilidade intermediária e resistência à vancomicina | |
| Helicobacter pylori, resistente à claritromicina | |
| Campylobacter spp., resistente às fluoroquinolonas | |
| Salmonella, resistente às fluoroquinolonas | |
| Neisseria gonorrhoeae, resistente à cefalosporina e às fluoroquinolonas | |
| Prioridade 03: Média | Streptococcus pneumoniae, sem sensibilidade à penicilina. |
| Haemophilus influenzae, resistente à ampicilina | |
| Shigella spp., resistente às fluoroquinolonas |
Adaptado de WHO, 2017.(10)
Quadro 2
Atalhos metodológicos
| Atalho metodológico | Descrição | Limitação |
| Tempo de publicação | Trabalhos publicados nos últimos 5 anos | Exclusão de artigos relevantes publicados há mais de 5 anos. |
| Consulta de literatura cinzenta | Sim | Baixo rigor metodológico |
| Idiomas de publicação | Português e inglês | Exclusão de artigos relevantes que foram publicados em outros idiomas. |
| Quantidade de repositórios científicos utilizados na busca | Dois repositórios | Exclusão de artigos relevantes publicados em outros repositórios. |
Fonte: Elaborado pelo autor (2023).
RESULTADOS E DISCUSSÃO
A resistência bacteriana é responsável por milhares de mortes em todo o mundo, onde o avanço de linhagens resistentes acaba por diminuir a eficácia da antibioticoterapia disponível atualmente.(13) No que tange a saúde única, é importante instigar a atuação multissetorial e multidisciplinar frente à RAM, uma vez que sua disseminação está diretamente relacionada a processos comuns de uma cadeia globalizada, como o trânsito de pessoas, comércio de animais vivos e de produtos alimentares, bem como por contato direto e/ou indireto, cadeia alimentar, água ou a partir do estrume utilizado na agricultura, conforme a Figura 1.(13,14)
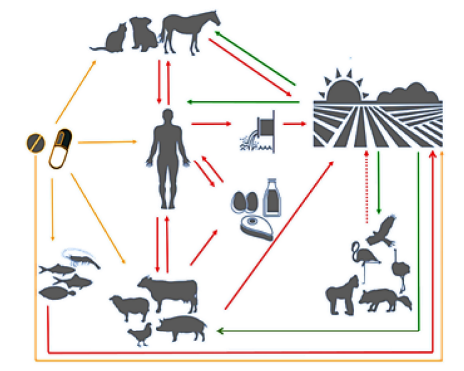
Figura 1
Vias de disseminação da resistência aos antimicrobianos
Fonte: Amaro; Correia; Clemente, 2020. (14)
Disseminação ambiental
Quando presentes no ambiente, os antimicrobianos mais persistentes, considerados estáveis, não são degradados pelos microrganismos presentes no solo, causando um desequilíbrio no ecossistema, e consequentemente contaminando o ambiente, fazendo com que tanto os ARG quanto as ARB sejam considerados poluentes emergentes.(7,15)
Diante da problemática da resistência bacteriana no ambiente, diversos fatores podem propiciar a disseminação desses genes e microrganismos, como a pobreza, controle ineficiente de infecções, escoamento de resíduos, ausência de saneamento básico e o tratamento inadequado – ou inexistente – da água, onde o lançamento de efluentes, bem como o lixo comum, aliado ao descarte incorreto dos medicamentos, pode contaminar não só o solo, mas também as águas de rios, lagos, oceanos e lençóis freáticos.(15,16)
As estações de tratamento de esgoto (ETE) recebem uma diversidade de resíduos, tendo em vista a composição das águas residuais, que podem ser oriundas do ambiente doméstico, clínico ou industrial, acumulando microrganismos e, consequentemente, seus genes de resistência.(17) Contudo, os tratamentos convencionais não permitem a remoção destes poluentes, favorecendo, assim, a disseminação da resistência aos antimicrobianos.(18,19)
Em consonância com essa informação, Abrantes e sua equipe (2022) realizaram um estudo em estações de tratamento de esgoto (ETE), onde detectaram bactérias tanto na entrada como na saída das estações, identificando 28 isolados de Aeromonas, incluindo as espécies A. hydrophila (16), A. caviae (9) e A. sobria (3), sendo resistentes a cefotaxima (100%), ciprofloxacino (75%), ceftriaxona (71%), ceftazidima (57%), cefoxitina (46%), cefepime (36%), piperacilina+tazobactam (28%) e gentamicina (11%).(20)
Ainda considerando os corpos aquáticos como importantes veículos, Scaccia e colaboradores (2022) coletaram a água da comunidade de São Remo, em São Paulo, para avaliar a presença de bactérias resistentes e seus genes correlacionados. Dos 67 isolados, 33 cepas foram positivas para os genes blaKPC (18,8%) e blaVIM (24,6%), sendo relacionados aos gêneros Aeromonas sp., Chryseobacterium sp., Elizabethkingia sp., Comamonas sp., Citrobacter sp., Escherichia sp., Pseudomonas sp., Enterobacter sp., Klebsiella sp., Kluyvera sp. e Serratia sp., apresentando resistência à cefotaxima (73%), amoxicilina (70%), aztreonam (54,5%) e ertapeném (51,5%).(21)
No estudo realizado pela equipe de Silva, em 2022, cepas de E. coli foram isoladas em diferentes pontos do Rio Carioca, as quais apresentaram alterações no perfil de sensibilidade aos antimicrobianos, principalmente em coletas posteriores à passagem do rio por unidades hospitalares. Neste trabalho, foram encontradas quatro cepas MDR, cuja resistência estava associada à ampicilina, ciprofloxacino, levofloxacino e sulfametoxazol-trimetoprim.(22)
Por fim, ressalta-se a possibilidade de a disseminação ambiental estar atrelada, ainda que indiretamente, à disseminação animal, uma vez que o uso de esterco animal na agricultura pode favorecer a propagação da resistência aos antimicrobianos, incluindo os genes de resistência à colistina (mcr-1) e sulfonamidas (sul1 e sul2).(23)
Disseminação animal
Como sugerido no estudo de Oliveira (2019),(23) animais também podem ser associados à disseminação de microrganismos e genes de resistência. Dentre os principais patógenos multirresistentes (MDR, do inglês, Multidrug resistant) que podem ser disseminados por animais, a Klebsiella pneumoniae tem sido citada como microrganismo de grande importância. Sousa e sua equipe (2019) identificaram 67 cepas de K. pneumoniae em animais domésticos (58,2%) e silvestres (41,8%), em que todos os isolados foram considerados multirresistentes, reforçando a ideia de que os animais podem ser considerados reservatórios de ARB.(1)
Todavia, outros microrganismos bacterianos também devem ser considerados de grande importância nesses estudos. Mariotini e Carvalho, em 2020, identificaram níveis elevados de resistência em isolados de cães, incluindo cepas de Staphylococcus spp. (44%), Bacillus spp. (8%), bactérias da ordem Enterobacterales (6%) e Pseudomonas spp. (3%). Além disso, os autores destacaram a presença de uma cepa de S. aureus resistente a todos os antibióticos testados, aumentando a preocupação quanto a disseminação destes microrganismos para o homem.(24)
Em 2022, Oliveira isolou cepas de E. coli produtoras de beta-lactamases de espectro estendido (ESBL, do inglês, Extended Spectrum Beta-Lactamase), oriundas de infecção do trato urinário em cães e gatos que foram atendidos no hospital veterinário da Universidade Federal Rural de Pernambuco.(25)
Calônico (2023), em sua revisão da literatura, discutiu a predominância de cepas de Staphylococcus sp. resistentes à meticilina, isolados de diferentes animais, como coelhos e lebres (64,52%), cães e gatos (30,62%), aves (22,41%), roedores (12,5%), suínos (5,55%), ruminantes (5%) e equinos (0,93%). Dentre as espécies isoladas, houve predominância de S. intermedius/pseudintermedius, S. aureus e S. schleiferi.(26)
Por sua vez, no estudo realizado por Melo (2023), 32 isolados de bactérias Gram-negativas oriundos de cães, gatos e coelhos foram testados, incluindo cepas de Escherichia coli (65,6%), Klebsiella pneumoniae (9,3%), Acinetobacter baumannii (6,3%), Pseudomonas aeruginosa (6,3%), Stenotrophomonas maltophilia (3,12%), Pseudomonas putida (3,12%), Acinetobacter pitti (3,12%) e Enterobacter hormaechei (3,12%), em que 62,5% foram ESBL positivos, além de serem resistentes a ceftriaxona (88%), ceftazidima (84%), cefepime (72%), ciprofloxacino (56%), tetraciclina (44%), cefoxitina (28%), amicacina (28%) e gentamicina (28%). Destes, 22 isolados foram considerados multirresistentes.(27)
Conforme supracitado, a poluição ambiental também pode estar diretamente relacionada à contaminação animal, como mostra o estudo de Castro (2023), onde sururus e ostras isolados de um mangue estavam contaminados com bactérias resistentes aos antimicrobianos. Dos 12 isolados, 8 foram considerados ESBL positivos e 4 apresentaram resistência aos aminoglicosídeos. (28) Além disso, a fim de manter a saúde do trato gastrointestinal de animais, como frangos de corte e suínos, a indústria agropecuária lança mão da adição de antibióticos na composição de rações, fato relatado desde a década de 1940.(29,30)
Muitos destes antimicrobianos, como a eritromicina, podem ser utilizados no tratamento humano, fazendo com que a resistência cruzada seja um perigo iminente. Bactérias resistentes, como Campylobacter jejuni e Campylobacter coli, podem ser facilmente transmitidos aos humanos a partir do consumo de carne contaminada, como mostra o estudo de Dias e colaboradores (2020), onde amostras do intestino de frangos de corte e de suínos foram utilizadas para investigar a resistência à eritromicina das estirpes de Campylobacter sp. isoladas. Todas as estirpes de C. coli de suínos (n=14) foram identificadas como resistentes e três, das 18 estirpes de C. jejuni isoladas de frangos, foram classificadas com resistência intermediária.(31)
Mendonça e sua equipe, em 2020, avaliaram o perfil de resistência de 51 isolados de Salmonella infantis, oriundos de amostras de frangos de corte. Cerca de onze perfis de resistência foram identificados, destacando-se amoxacilina (35,3%) e sulfonamida (15,7%), ainda que nenhum tenha sido considerado multirresistente.(32)
Por sua vez, Schmiedt (2021) caracterizou o perfil de resistência de isolados de Escherichia coli obtidos de amostras da cadeia produtiva de frangos de corte. Dos 701 isolados, 578 foram considerados MDR e apenas 15 (2,2%) foram ESBL positivo. Destes, foram detectadas taxas elevadas de resistência à ciprofloxacina (94,1%), tetraciclina (56,3%), sulfametoxazol+trimetroprim (54,3%) e amoxacilina (48,1%).(33)
Tais resultados são extremamente preocupantes, uma vez que tais antimicrobianos podem ser utilizados para tratamento de infecções em humanos, trazendo um alerta quanto ao perigo para infecções alimentares, visto que estes animais podem ser consumidos, originando uma nova via de disseminação da RAM.
Disseminação alimentar
Há uma preocupação constante com as doenças transmitidas por alimentos, uma vez que diversos agentes biológicos podem ser veiculados desta forma. Um dos maiores exemplos é a Salmonelose, comumente associada ao consumo de carne de frango, ovos e derivados.(34,35) Por conseguinte, foram identificados 23 sorotipos de Salmonella resistentes aos antimicrobianos, incluindo o Florfenicol, um dos principais antibióticos utilizados na produção animal.(36)
Embutidos cárneos, como salames e linguiças coloniais, também podem ser grandes fontes de contaminação, como mostra o estudo de Henning (2019), onde embutidos cárneos fermentados produzidos em Francisco Beltrão, no Paraná, estavam contaminados por E. coli (39%), Salmonella spp. (61%) e Staphylococcus coagulase positiva (39%). Destes isolados, cerca de 16% não apresentaram resistência aos antimicrobianos testados, enquanto os demais (84%) apresentaram resistência a pelo menos um dos antimicrobianos, incluindo cepas multirresistentes de Salmonella spp.(37)
Sousa, em 2020, isolou e identificou o perfil de resistência de microrganismos provenientes de ovos de galinha caipira comercializados em feiras livres no semiárido brasileiro. Neste estudo, 31,3% dos ovos apresentaram crescimento bacteriano, com isolados de Staphylococcus spp. (27,5%), Bacillus spp. (15%), Pseudomonas aeruginosa (7,5%), Klebsiella spp. (7,5%), Salmonella spp. (7,5%), Proteus mirabilis (7,5%), Citrobacter spp. (7,5%), Escherichia coli (7,5%), Providencia spp. (5%), Corynebacterium spp. (2,5%), Enterobacter spp. (2,5%) e Alcaligens spp. (2,5%).(38)
Além disso, mais da metade das cepas Gram-negativas foram positivas para ESBL, em que genes de beta-lactamases, como o CTX-M, foram detectados em quatro isolados, sendo um isolado de Klebsiella spp. apresentando o grupo blaCTX-M2-like e três cepas (Klebsiella spp., Salmonella spp. e Citrobacter spp.) que apresentaram o gene blaCTX-M8-like.(38)
Isolados de Salmonella spp. também foram identificados em amostras de filé de tilápia fresca comercializadas no Distrito Federal. Os genes de resistência blaCTX (64,9%), tetB (54,4%), floR (50,9%) e sul2 (49,1%) foram identificados, corroborando para com as taxas de resistência identificadas para amoxicilina/ácido clavulânico (87,7%), tetraciclina (82,5%), sulfonamidas (57,9%) e cloranfenicol (26,3%), em que 56,1% das cepas isoladas foram consideradas multirresistentes.(39)
Contudo, tais microrganismos também podem ser encontrados como parte da microbiota intestinal dos animais, como mostra o estudo realizado por Sampaio e Motta (2021), onde 50 peixes da espécie Oncorhynchus mykiss foram coletados, assim como 9 amostras de água dos tanques ativos, obtendo 222 isolados bacterianos, incluindo espécies da Ordem Enterobacterales, famílias Aeromonaceae e Pseudomonadaceae; além de bactérias Gram positivas, como os gêneros Staphylococcus sp., Enterococcus sp., Lactococcus sp., Streptococcus sp., Kocuria spp. e Gemella spp.(40)
Os autores relatam que o perfil de resistência a, pelo menos, um antimicrobiano foi observado em 65,3% (n=79) das bactérias Gram-negativas e 82,2% (n=83) das Gram-positivas, enquanto o perfil multirresistente foi observado em 7,4% (n=9) e 36,6% (n=37), respectivamente.(40)
Por sua vez, no estudo realizado por Oliveira e sua equipe (2021), cepas de Salmonella sp. e de E. coli foram isoladas de água de coco comercializada no norte mato-grossense, em que todas as cepas de E. coli foram resistentes a cefalotina, norfloxacina e ciprofloxacina. Dessas, duas foram resistentes à amoxicilina e ao ácido clavulânico e uma à ampicilina, sendo consideradas multirresistentes. Em contrapartida, apenas uma cepa de Salmonella foi considerada MDR, apresentando resistência à ampicilina, cefalotina e amoxicilina + ácido clavulânico.(41)
Outro ponto que merece destaque é a possibilidade de contaminação cruzada, onde ocorre a transferência de microrganismos de um material ou alimento contaminado para outro, como é o caso das superfícies de cortes, utensílios amplamente utilizados no preparo e manipulação de alimentos crus ou prontos para o consumo.(42)
Os achados de Teodoro (2021) corroboram para com a afirmação supracitada, uma vez que espécies de A. hydrophila (20,83%), A. caviae (26,39%) e A. veronii (44,44%) foram identificadas em amostras de temakis. Cerca de 90% dos isolados apresentaram resistência à amoxicilina-ácido clavulânico, 17% à tetraciclina, 10% ao imipeném e 3% ao aztreonam.(43)
Por fim, ressalta-se que a utilização de antibióticos para fins profiláticos, terapêuticos e agropecuários avança em ritmo constante, e que muitos fármacos utilizados no âmbito veterinário ou agropecuário também podem ser empregados no tratamento de infecções em humanos, favorecendo ainda mais a resistência cruzada e, consequentemente, a disseminação de microrganismos resistentes e seus genes relacionados.
CONSIDERAÇÕES FINAIS
A disseminação de ARG e ARB se torna cada vez mais frequente em diferentes vias, estando intimamente relacionada à Saúde única, facilitando a troca de material genético entre bactérias ditas ambientais para com as patogênicas e, consequentemente, permitindo a disseminação desses genes de resistência. Dessa maneira, os resultados apresentados apontam o potencial risco de disseminação da RAM por diferentes fontes.
Com base em tudo que foi descrito no estudo, acredita-se que existe uma necessidade de aprimorar a vigilância ambiental, além de estabelecer um controle mais rigoroso quanto ao uso de antimicrobianos, a fim de tentar mitigar o avanço da resistência bacteriana, tendo em vista o risco elevado para a saúde pública mundial.
REFERÊNCIAS
- Sousa ATHI, et al. Perfil de resistência antimicrobiana de Klebsiella pneumoniae isoladas de animais domésticos e silvestres. Arquivo Brasileiro de Medicina Veterinária e Zootecnia, v. 71, p. 584-593, 2019.
- Brito GB, Trevisan M. O uso indevido de antibióticos e o eminente risco de resistência bacteriana. Revista Artigos. Com, v. 30, p. e7902-e7902, 2021.
- Teixeira AR, et al. Resistência bacteriana relacionada ao uso indiscriminado de antibióticos. Saúde em Foco, n. 11, 2019.
- Silva LOP. Aspectos gerais da COVID-19 e as possíveis consequências do uso indiscriminado de antimicrobianos durante a pandemia no Brasil. 116f. Dissertação (Mestrado em Saúde Pública e Meio Ambiente), Escola Nacional de Saúde Pública Sérgio Arouca, Fundação Oswaldo Cruz, Rio de Janeiro, 2023.
- WHO Report on Surveillance of Antibiotic Consumption. World Health Organization, 128p, 2018.
- Portela G. Fiocruz no Ar – Uso de antibióticos e nutrição: evitando excessos e se cuidando. 2020. Disponível em: https://portal.fiocruz.br/noticia/fiocruz-no-ar-uso-de-antibioticos-e-nutricao-evitando-excessos-e-se-cuidando#:~:text=O%20pa%C3%ADs%20est%C3%A1%20em%2017%C2%BA,consumo%20de%2018%20doses%20di%C3%A1rias. Acesso em: 12 set. 2023.
- Reis AC, et al. Biodegradation of antibiotics: The new resistance determinants – part I. New biotechnology, v. 54, p. 34-51, 2020.
- O’Neill J. Antimicrobial Resistance: Tackling a crisis for the health and wealth of nations. The Review on Antimicrobial Resistance, 20p, 2014.
- Silva LOP, Nogueira JMR. Resistência bacteriana: potencial de plantas medicinais como alternativa para antimicrobianos. Revista Brasileira de Análises Clínicas, v. 23, n. 1, p. 21-27, 2021.
- WHO publishes list of bacteria for which new antibiotics are urgently needed. 2017. Disponível em: https://www.who.int/news/item/27-02-2017-who-publishes-list-of-bacteria-for-which-new-antibiotics-are-urgently-needed. Acesso em: 14 set. 2023.
- Souza JBB, et al. Uso de antimicrobianos na pecuária e a resistência microbiana em humanos. In: Congresso Interdisciplinar. 2022.
- Jadeja NB, Worrich A. From gut to mud: dissemination of antimicrobial resistance between animal and agricultural niches. Environmental Microbiology, v. 24, n. 8, p. 3290-3306, 2022.
- OPAS/OMS. Relatório sinaliza aumento da resistência a antibióticos em infecções bacterianas em humanos. 2022. Disponível em: https://www.paho.org/pt/noticias/9-12-2022-relatorio-sinaliza-aumento-da-resistencia-antibioticos-em-infeccoes-bacterianas. Acesso em: 14 set. 2023.
- Amaro A, Correia I, Clemente L. Resistência aos antibióticos em bactérias com origem em animais da cadeia alimentar. 2020. Disponível em: https://www.veterinaria-atual.pt/destaques/resistencia-aos-antibioticos-em-bacterias-com-origem-em-animais-da-cadeia-alimentar/. Acesso em: 14 set. 2023.
- Silva LC, Cardoso A, Vieira JMBD. Dispersão da resistência a antimicrobianos no ambiente sob o conceito de Saúde Única. Concilium, v. 22, n. 6, p. 937-948, 2022.
- Pinto GMF, et al. Estudo do descarte residencial de medicamentos vencidos na região de Paulínia (SP), Brasil. Engenharia Sanitária e Ambiental, v. 19, p. 219-224, 2014.
- Joseph SM, et al. Longitudinal comparison of bacterial diversity and antibiotic resistance genes in New York City sewage. Msystems, v. 4, n. 4, 2019.
- Silva TCB. Participação das estações de tratamento de esgoto na disseminação de genes de resistência. 57f. Trabalho de conclusão de curso (Graduação em Biomedicina), Instituto de Ciências Básicas da Saúde, Universidade Federal do Rio Grande do Sul, 2020.
- Silva LOP, Abrantes JÁ, Nogueira JMR. Monitoramento de efluentes hospitalares como ferramenta para a vigilância de patógenos multirresistentes. Peer Review, v. 5, n. 19, p. 1-11, 2023.
- Abrantes JÁ, et al. Identificação e perfil de susceptibilidade de Aeromonas isoladas de Estações de Tratamento de Esgoto utilizando meio cromogênico com antimicrobiano. Brazilian Journal of Development, v.8, n.5, p.38227-38240, 2022.
- Scaccia N, et al. Disseminação ambiental de resistência a antibióticos através dos aglomerados subnormais. The Brazilian Journal of Infectious Diseases, v. 26, p. 102401, 2022.
- Silva TSM, et al. Perfil de sensibilidade aos antimicrobianos das cepas de Escherichia coli isoladas de amostras de águas superficiais do Rio Carioca-RJ, Brasil. Engenharia Sanitaria e Ambiental, v. 27, p. 673-682, 2022.
- Oliveira CC. Ocorrência de Genes de Resistência a Antimicrobianos em Solos de Áreas Agrícolas e de Reserva Legal em Nova Friburgo, RJ. 60p. Dissertação (Mestrado em Agronomia – Ciência do Solo), Universidade Federal Rural do Rio de Janeiro, 2019.
- Mariotini AB, Carvalho EV. Perfil de Resistência aos Antibióticos de bactérias isoladas de infecções de animais atendidos no UNIFAA. Revista Saber Digital, v. 13, n. 1, p. 176-187, 2020.
- Oliveira LBS. Isolamento e detecção de bactérias produtoras de β-lactamases de espectro estendido (ESBL) oriundos de infecção do trato urinário em cães e gatos atendidos no hospital veterinário da Universidade Federal Rural de Pernambuco. 43p. Trabalho de conclusão de curso (Especialização em Medicina Veterinária Preventiva), Universidade Federal Rural de Pernambuco, 2022.
- Calônico VS. Espécies de Staphylococcus resistentes à meticilina isoladas de animais. 45p. Trabalho de conclusão de curso (Graduação em Farmácia), Universidade Federal de Santa Catarina, 2023.
- Melo JG. Bactérias Gram-negativas colonizadoras do trato gastrointestinal de animais domésticos e seus fenótipos de resistência aos beta-lactâmicos. 70p. Trabalho de conclusão de curso (Graduação em Farmácia), Universidade Federal de Santa Catarina, 2023.
- Castro LH. Investigação de bactérias Gram-negativas multirresistentes de importância clínica em bivalves marinhos isolados de mangue, na cidade de Vitória, Espírito Santo. 36p. Trabalho de conclusão de curso (Graduação em Biomedicina), Instituto Federal do Espírito Santo, Vila Velha, 2023.
- Reis TL, Vieites FM. Antibiótico, prebiótico, probiótico e simbiótico em rações de frangos de corte e galinhas poedeiras. Ciência Animal, v. 29, n. 3, p. 133-147, 2019.
- Molinari PV, et al. Alternativas ao uso de antibióticos via ração na produção de suínos. Anais da Mostra Nacional de Iniciação Científica e Tecnológica Interdisciplinar (MICTI)-e-ISSN 2316-7165, v. 1, n. 13, 2020.
- Dias TS, et al. Phenotypic and molecular characterization of erythromycin resistance in Campylobacter jejuni and Campylobacter coli strains isolated from swine and broiler chickens. Pesquisa Veterinária Brasileira, v. 40, p. 598-603, 2020.
- Mendonça EP, et al. Characteristics of virulence, resistance and genetic diversity of strains of Salmonella Infantis isolated from broiler chicken in Brazil. Pesquisa Veterinária Brasileira, v. 40, p. 29-38, 2020.
- Schmiedt JA. Caracterização do perfil fenotípico e genotípico de resistência a antimicrobianos de isolados de Escherichia coli em uma cadeia produtiva de frango de corte. Dissertação (Mestrado em Ciência Animal), Universidade Federal do Paraná, 2021.
- Silva EM, Duarte A. Salmonella Enteritidis em aves: retrospectiva no Brasil. Brazilian Journal of Poultry Science, v. 4, p. 85-100, 2002.
- Hoelzer K, Moreno Switt AI, Wiedmann M. Animal contact as a source of human non-typhoidal salmonellosis. Veterinary research, v. 42, n. 1, p. 1-28, 2011.
- Ansiliero R, Gelinski JLN, Scheffmacher MGC. Identificação e avaliação da susceptibilidade a antimicrobianos de sorotipos de salmonella sp. De uma cadeia produtiva de frangos de corte do Sul do Brasil. Evidência, v. 19, n. 1, p. 57-72, 2019.
- Henning K. Embutidos cárneos fermentados produzidos em Francisco Beltrão, Paraná: avaliação físico-química, microbiológica e perfil de resistência bacteriana. 62f. Dissertação (Mestrado em Ciências Aplicadas à Saúde), Universidade Estadual do Oeste do Paraná, 2019.
- Sousa DLC. Perfil de resistência antimicrobiana de bactérias isoladas de ovos de galinhas caipiras (Gallus Gallus domesticus) no Semiárido do Nordeste do Brasil e sua aplicação em saúde única. 53f. Dissertação (Mestrado em Ciência Animal), Universidade Federal de Campina Grande, 2020.
- Ferreira ACAO. Ocorrência de Salmonella em tilápia fresca comercializada no Distrito Federal e avaliação do perfil de resistência antimicrobiana das cepas isoladas. 89f. Tese (Doutorado em Ciências e Tecnologias em saúde), Universidade de Brasília, 2021.
- Sampaio NB, Motta OV. Análise da microbiota intestinal de truta arco-íris (Oncorhynchus mykiss, WALBAUM) e seu ambiente aquático na Região Serrana do Estado do Rio de Janeiro. In: Congresso Fluminense de Pós-Graduação – CONPG. 2021.
- Oliveira SMP, et al. Perfil de sensibilidade antimicrobiana de Escherichia coli e Salmonella isolados de água de coco “in natura” comercializadas no norte-mato-grossense. Scientific Electronic Archives, v. 14, n. 12, 2021.
- Oliveira JP, Silva DC, Pereira JP. Contaminação cruzada: Uso de tábuas de corte na manipulação de alimentos no ambiente doméstico. Pubvet, v. 17, n. 04, p. e1380, 2023.
- Teodoro JR. Potencial patogênico e susceptibilidade aos antimicrobianos de Aeromonas em alimentos prontos para o consumo. 95p. Dissertação (Mestrado em Engenharia de Alimentos), Universidade Estadual de Campinas, 2021.
Correspondência
Líllian Oliveira Pereira da Silva
E-mail: [email protected]
