Análise do perfil de sensibilidade aos antimicrobianos de um hospital público de Aracaju, Sergipe
Antimicrobial sensitivity profile analysis of a public hospital in Aracaju, Sergipe
Henrique Francisco de Almeida1
Roberta Oliveira da Silva2
Mônica Batista de Almeida3
Flávia de Almeida4
Jéssica Santos Menezes5
Thays Almeida Pereira Dias6
1 Programa de Pós-graduação Strictu sensu em Biologia Parasitária da Universidade Federal de Sergipe.
2 Graduada em Farmácia pela Universidade Tiradentes.
3 Doutora em Biotecnologia pela Universidade Federal de Sergipe.
4 Graduada em Farmácia pela Universidade Federal de Sergipe.
5 Graduanda em Enfermagem pela Faculdade Estácio de Sergipe.
6 Graduada em Biomedicina pela Universidade Paulista.
Instituição: Universidade Tiradentes. Aracaju – SE, Brasil.
Recebido em 14/09/2020
Aprovado em 07/05/2021
DOI: 10.21877/2448-3877.202102066
INTRODUÇÃO
Os antibióticos são medicamentos que são utilizados como recursos terapêuticos para tratar doenças infecciosas provocadas por microrganismos e, dessa maneira, diminuem os índices de morbimortalidade relacionados às infecções causadas por bactérias.(1) A resistência a esses antimicrobianos disponíveis para a terapêutica é um problema que cresce em ritmo acelerado no mundo. Esse fenômeno ocorre devido à ineficácia de antibióticos que são comumente empregados para microrganismos outrora sensíveis e que, agora, tornaram-se resistentes à ação desses medicamentos.(2)
A resistência microbiana acontece de forma natural mediante a herança genética de plasmídeos, por meio da pressão seletiva provocada pelo antibiótico quando as bactérias suscetíveis são eliminadas, ficando apenas as resistentes, as quais passam a se reproduzir, e, também, por intermédio de mutações.(3,4)
No entanto, há fatores que agilizam esse processo, a saber: o uso inadequado de antimicrobianos nos seus mais variados âmbitos, seja pelos profissionais de saúde, pela sociedade ou em animais, prática essa amplamente utilizada.(5) Um dos ambientes onde a resistência antimicrobiana propaga-se com intensa rapidez é o hospitalar.(6) Esse centro de saúde é um local propício para a ocorrência de infecções por microrganismos resistentes e multirresistentes, primeiramente, porque é um ambiente com uma grande variedade de micróbios e, também, porque é neste âmbito que se encontra um considerável contingente de pacientes com enfermidades, tornando-os vulneráveis por causa da sua situação clínica, e por, na maioria dos casos, serem submetidos a diversos procedimentos invasivos, o que o faz entrar em um quadro de debilidade imunológica e, assim, mais suscetível a adquirir infecções.(7)
A equipe de saúde empenhada em tratar os pacientes com quadros infecciosos utiliza-se de antimicrobianos. Por muitas vezes, esses medicamentos são usados de forma irracional, seja pelo uso de maneira excessiva ou pela ausência de necessidade de tratamento com esses fármacos.(8,9) O uso indiscriminado de antibióticos promove o aumento e a disseminação da resistência bacteriana, o que torna mais difícil a intervenção nas doenças infecciosas, devido à ausência de medicamentos que tratem essas enfermidades.(10,11)
Sendo assim, entende-se, também, que a dificuldade em tratar as infecções interfere diretamente na duração do tempo de hospitalização, o que eleva os custos hospitalares, reduz as opções terapêuticas eficazes, bem como aumenta as taxas de mortalidade dos pacientes internados.(6-12) Além do uso irracional de medicamentos utilizados no combate às enfermidades bacterianas, outra forma de propagação da resistência microbiana, no contexto hospitalar, é por meio das Infecções Relacionadas à Assistência à Saúde (IRAS),(13,14) cujo termo é uma substituição e uma ampliação do conceito de Infecção Hospitalar (IH).(15)
As IRAS são adquiridas, dentre outras maneiras, através da contaminação cruzada que é uma forma de disseminação de microrganismos patogênicos. Essa transmissão está relacionada aos pertences e equipamentos que estão em volta de indivíduos colonizados, os quais servem de reservatório para as bactérias.(16) O interesse por esse tema surgiu devido à necessidade de conscientização por parte dos profissionais de saúde e da população quanto ao uso racional de antimicrobianos, visto que a utilização indiscriminada destes eleva os índices de resistência bacteriana. Neste sentido, a presente pesquisa tem como objetivo identificar quais são as principais bactérias causadoras de infecção e qual a incidência desses patógenos, buscando avaliar o perfil de sensibilidade desses microrganismos em um hospital público de Aracaju – Sergipe.
MATERIAL E MÉTODOS
Trata-se de um estudo retrospectivo, transversal, realizado em um hospital público localizado em Aracaju – Sergipe. A coleta secundária dos dados foi realizada mediante a autorização da administração da instituição hospitalar, conforme o termo de compromisso e aceite de realização da pesquisa concedido pela Coordenação do Serviço de Educação Continuada do hospital. Foram coletados e analisados os registros e relatórios de cepas bacterianas e de alguns fungos provenientes de pacientes internados nas Unidades de Terapia Intensiva (UTIs) neonatal, pediátrica e adulto, que realizaram culturas de material biológico com o teste de sensibilidade a antimicrobianos (TSA), no período compreendido entre janeiro de 2017 a dezembro de 2018. Esses materiais foram fornecidos pela Comissão de Controle de Infecção Hospitalar (CCIH), que possuía esses dados devidamente registrados. Durante a realização da coleta de dados não foi necessário ter acesso aos dados dos pacientes internados, por isso não foi preciso submeter este estudo ao comitê de ética.
Portanto, não foi necessário aplicar o termo de consentimento livre e esclarecido. É mister ressaltar que esse método está de acordo com os estudos realizados por Bordignon e Lima(7) e Santos et al.,(4) os quais desenvolveram o mesmo tipo de pesquisa em um hospital do sudoeste do Paraná e em outra instituição localizada em Macapá, no estado do Amapá, respectivamente. Para a tabulação dos resultados, foi utilizado o software Excel 2018, em que foram utilizados gráficos, nos quais contêm os principais microrganismos isolados, a sua incidência e os perfis de sensibilidade e resistência aos antimicrobianos. Os dados foram analisados pela estatística descritiva.
RESULTADOS
Primeiramente, foram coletados e analisados os perfis microbiológicos e de sensibilidade dos anos de 2017 e 2018 provenientes das Unidades de Terapia Intensiva (UTIs) neonatal, pediátrica e adulto. No perfil microbiológico da UTI Neonatal de 2017 foram isolados quatro tipos de bactérias, com prevalência de espécies de Enterobacter spp., com seis ocorrências (46,1%), seguidos de Escherichia coli com quatro achados (30,8%), Pseudomonas spp. com duas ocorrências (15,4%) e Serratia spp. com uma ocorrência (7,7%). Em 2018, o número de espécies isoladas evoluiu para 17 tipos. Houve elevadas taxas de infecções por Staphylococcus coagulase negativa (SCN), no total de 18 isolados (30%), S. aureus e Staphylococcus aureus resistente à meticilina (MRSA) com nove e três achados, respectivamente, o que equivale, no total, a 20% dos casos, Escherichia coli e Klebsiella pneumoniae b-lactamase de Espectro Estendido (ESBL) com cinco ocorrências cada (totalizando 18%), Enterococcus spp., Enterobacter spp. ESBL, P. aeruginosa, Enterobactéria Resistente aos Carbapenêmicos (ERC) e Leveduras com três casos cada (no total de 20%), Listeria monocytogenes , Serratia (spp. e marcescens), com duas ocorrências cada (no total de 6%) e, por fim, Morganella morganii, Streptococcus agalactiae e Providencia stuartii ESBL, com um caso cada (totalizando 6%).
No perfil microbiológico da UTI Pediátrica do ano de 2017 foram isolados seis tipos de microrganismos, com destaque na prevalência de cepas pertencentes às enterobactérias, no total de quatro ocorrências (30,8%), seguidos de Pseudomonas aeruginosa, com dois achados (15,4%), Serratia spp., Pseudomonas spp. e Proteus mirabilis com uma ocorrência (23%). Foram isolados também fungos do gênero Candida spp. (quatro casos, 30,8%).
Já no ano de 2018 houve uma mudança no quadro de ocorrências. Houve, no total, 61 achados, sendo que prevaleceu casos de infecções por P. aeruginosa (22 casos, o que corresponde a 36%), seguidos de Staphylococcus coagulase negativa (sete casos, 11,7%), Staphylococcus aureus e Escherichia coli com seis casos cada (19,7%), Enterobacter cloacae (três casos, 4,9%) Klebsiella spp. e Klebsiella pneumoniae com uma e duas ocorrências, respectivamente, o que equivale a 4,9% dos achados; as espécies de Acinetobacter spp., Serratia spp., Burkholderia cepacea, Stenotrophomonas maltophilia correspondem a 6,5% das ocorrências, equivalente a um caso cada microrganismo. Foram isoladas espécies de Candida spp. (seis achados, 9,8 %) e Leveduras (quatro ocorrências, 6,5%).
No perfil microbiológico da UTI Adulto do ano de 2017 foram isolados quatro tipos de microrganismos, são eles: Enterobacter spp. com quatro ocorrências (57%), Pseudomonas spp., Serratia spp. e Enterococcus spp. com um achado cada (no total de 43%). Já em 2018 foram isoladas 10 tipos com destaque para a prevalência de infecções por Escherichia coli e P. aeruginosa com sete ocorrências cada (no total de 43,7%), Acinetobacter spp. com cinco achados clínicos (15,6%), S. aureus com quatro ocorrências (12,5%), Enterococcus spp. com três casos (9,4%), Klebsiella oxytoca com duas ocorrências (6,3%), Pseudomonas spp., SCN, Candida spp. e Leveduras com um achado cada (totalizando 12,5%). A Tabela 1 traz a incidência dos microrganismos isolados nas UTIs Neonatal, Pediátrica e Adulto dos anos de 2017 e 2018.
Os testes de sensibilidade aos antimicrobianos (TSA) foram realizados para os microrganismos mais incidentes em cada um dos setores envolvidos no estudo no período de 2017 e 2018. Os resultados dos testes da UTI Neonatal no ano de 2017 para as cepas das enterobactérias evidenciaram algumas classes de antibióticos que mostraram sensibilidade igual e/ou inferior a 50%, sendo eles: amicacina e ciprofloxacino (50%), aztreonam (40%), ceftriaxona e cefotaxima (33,33%), cefalotina e cefepime (20%) e gentamicina (16,67%). Apenas para a associação entre piperacilina+tazobactam, as bactérias apresentaram 100% de sensibilidade, seguidos de imipenem e meropenem (83,33%) e sulfametoxazol+trimetoprim (60%), enquanto que se apresentaram 100% resistentes à associação entre amoxicilina+clavulanato.
O perfil de sensibilidade das Escherichia coli demonstra que elas apresentaram sensibilidade igual e/ou inferior aos seguintes antimicrobianos: sulfametoxazol+trimetoprim, ceftazidima e aztreonam (50%), cefalotina (33,33%), enquanto que se mostraram totalmente sensíveis a amicacina, ciprofloxacino, nitrofurantoína, meropenem e imipenem, seguidos de ceftriaxona, cefepime, cefotaxima e amoxicilina+clavulanato (66,67%).
Para o ano de 2018, foram realizados TSA para Staphylococcus coagulase negativa (SCN), S. aureus e MRSA. Os antibióticos utilizados nos testes e as porcentagens de sensibilidade para SCN estão descritos a seguir: os antimicrobianos que apresentaram sensibilidade nas culturas igual e/ou inferior a 50% foram: ciprofloxacino, clindamicina, sulfametoxazol+trimetoprim (50%), tetraciclina (33%), ampicilina+sulbactam, cefepime, cefoxitina, meropenem, oxacilina e piperacilina+tazobactam (25%). Essa cepa foi totalmente resistente a ampicilina, azitromicina, cefaclor, penicilina G, cefazolina, eritromicina e gentamicina.
O perfil de sensibilidade para S. aureus e MRSA demonstra que elas apresentaram total sensibilidade para azitromicina, cefaclor, cefazolina, linezolida, rifampicina e tetraciclina, seguidos por, em ordem decrescente, para clindamicina, eritromicina, sulfametoxazol+trimetoprim (40%) e gentamicina (20%). Esses microrganismos foram totalmente resistentes à ampicilina e à penicilina G.
Na UTI Pediátrica, os resultados do TSA de 2017 para as cepas das enterobactérias evidenciaram algumas classes de antibióticos que mostraram sensibilidade igual e/ou inferior a 50%, sendo eles: gentamicina (50%), ceftriaxona/cefotaxima e cefepima (25%). Amicacina e meropenem foram os únicos antimicrobianos que apresentaram 100% de sensibilidade nas culturas, seguidos de ciprofloxacino (75%).
O perfil de sensibilidade das P. aeruginosa revela que as cepas mostraram uma sensibilidade de 50% para amicacina/gentamicina, enquanto que se apresentaram 100% resistentes ao ciprofloxacino. Para o ano de 2018, foram realizados TSA para as espécies de P. aeruginosa e S. aureus. Os antibióticos utilizados nos testes e as porcentagens de sensibilidade da P. aeruginosa estão descritos a seguir: amicacina e gentamicina (64%), cefepima (36%), piperacilina+tazobactam (82%), imipenem/meropenem e ciprofloxacino (63%). A cepa demonstrou sensibilidade de 100% apenas para colistina (polimixina E) e a polimixina B. A espécie de S. aureus mostrou 100% de resistência para ampicilina, ampicilina+sulbactam e 50% para clindamicina, enquanto que para vancomicina, teicoplanina e linezolida foram totalmente sensíveis.
O perfil de sensibilidade para o ano de 2017 na UTI Adulto revelou que a Enterobacter spp. teve sensibilidade para ceftriaxona/cefotaxima (33%), cefepima e gentamicina (50%) amicacina e ciprofloxacino (67%) e meropenem (80%). Já o perfil de sensibilidade de 2018 foi realizado unicamente para as cepas de Acinetobacter spp., sendo sensíveis para os seguintes antimicrobianos: gentamicina (50%), amicacina (33,33%), cefepima, piperacilina+tazobactam, ciprofloxacino e meropenem (25%). A única droga que as bactérias mostraram-se totalmente sensíveis foi a colistina. Para fins de ajuste na Tabela 2, a qual mostra a porcentagem de sensibilidade aos antimicrobianos utilizados no Teste de Suscetibilidade aos Antibióticos para cada espécie de bactérias, ficou definido que os nomes das bactérias serão descritos da seguinte forma:
Enterobacter spp: X1;
Escherichia coli: X2;
Staphylococcus coagulase negativa: X3;
Staphylococcus aureus: X4;
Staphylococcus aureus resistente à meticilina: X5;
Pseudomonas aeruginosa: X6;
Acinetobacter spp.: X7.
Tabela 1 – Incidência dos microrganismos isolados nas UTIs Neonatal, Pediátrica e Adulto dos anos 2017 e 2018
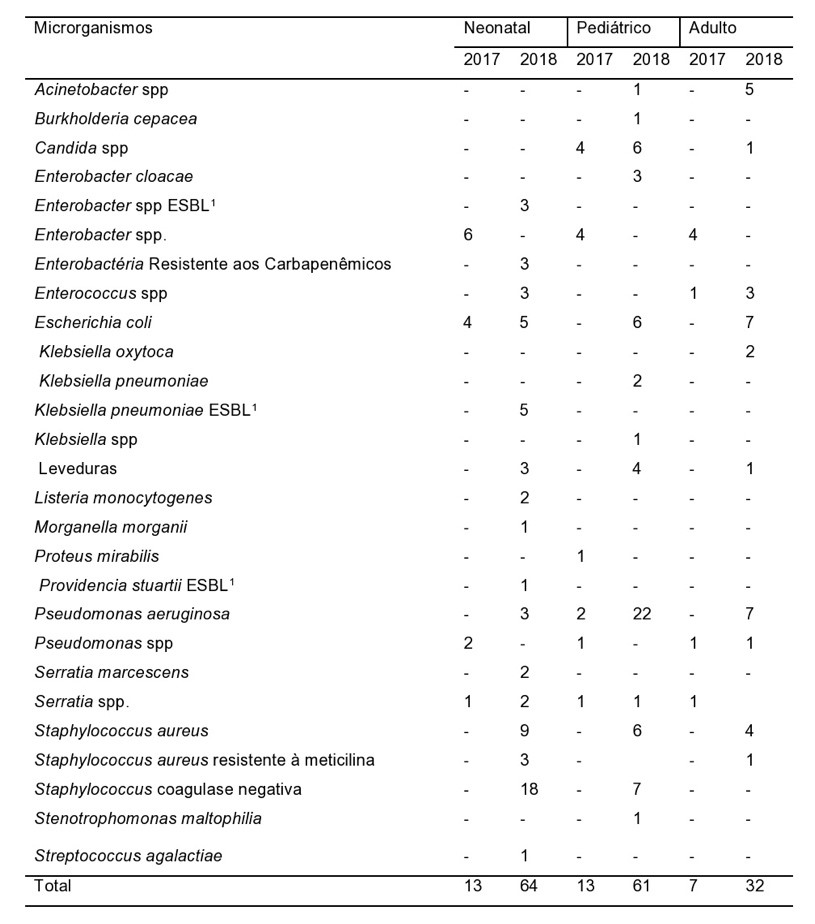
*β-lactamase de espectro estendido1
Fonte: Dados da pesquisa.
Tabela 2 – Porcentagem de sensibilidade dos antibióticos utilizados no TSA para cada espécie de bactérias
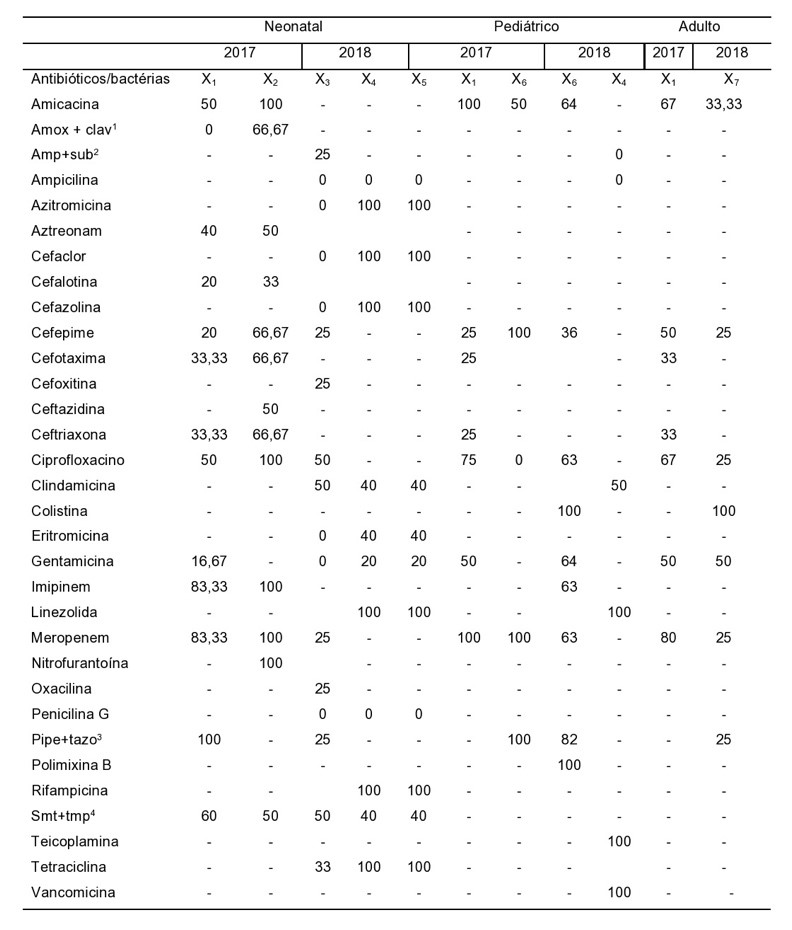
*Amoxicilina+Clavulanato1, Ampicilina+Sulbactam2, Piperacilina+Tazobactam3, Sulfametoxazol+Trimetoprim4. Entende-se 0 referente a 100% de resistência.
DISCUSSÃO
De acordo com os autores Blair et al.(17) e Lima et al.,(18) as bactérias desenvolvem e utilizam-se de vários mecanismos de sobrevivência, a fim de resistirem à ação das drogas. Alguns desses mecanismos de resistência foram observados neste estudo, como Staphylococcus aureus MRSA (três casos), Klebsiella pneumoniae ESBL (cinco casos), Enterobacter spp ESBL (três casos), P. aeruginosa ERC (três casos) e Providencia stuartii ESBL (1 caso), todas elas presentes na UTI neonatal, corroborando com os estudos de Cadot et al.(19) e Bowen et al.,(20) que demonstram que os recém-nascidos são mais vulneráveis às infecções por bactérias multirresistentes, devido à sua imaturidade biológica. É importante ressaltar que conforme um estudo realizado, foi evidenciado que a Pseudomonas aeruginosa é o principal patógeno causador de pneumonia associada à ventilação mecânica (PAVM) em pacientes críticos internados na UTI. (21)
As bactérias que produzem ESBL são capazes de hidrolisar o anel b-lactâmico de todas as classes de antimicrobianos que expressem essa estrutura, por exemplo, as penicilinas, as cefalosporinas e monobactâmicos.(7,22)
A Figura 1 traz os nomes das espécies de cepas multirresistentes que foram isoladas na UTI neonatal.
No estudo conduzido por Bordignon e Lima,(7) bem como o presente trabalho, constatou-se que as enterobactérias têm elevadas taxas de resistência aos antibióticos b-lactâmicos, enquanto que o MRSA apresentou sensibilidade a alguns antimicrobianos dessa classe terapêutica, o que diverge desses autores, que, em seu trabalho, relataram total resistência desse microrganismo a esses fármacos.
Os trabalhos realizados por Almeida et al.(23) e Andrade et al.(24) evidenciaram que as bactérias pertencentes à família Enterobacteriaceae são as que produzem mais comumente as b-lactamases de espectro estendido (ESBL), ou seja, favorecendo a resistência desses microrganismos aos antimicrobianos que possuem o anel b-lactâmico. De acordo com os estudos desenvolvidos por esses autores, foi constatado que as enterobactérias desenvolveram esse tipo de mecanismo de defesa predominantemente em hospitais e que esses patógenos estão relacionados, também, a infecções alimentares através do consumo de frutos do mar (peixes e camarões).
A Tabela 3 mostra a média geral do perfil de sensibilidade para os microrganismos isolados.
Por fim, os antibióticos que demonstraram menores índices nos perfis de sensibilidade na média geral para os microrganismos isolados foram: gentamicina, ampicilina e amoxicilina+clavulanato (utilizados para enterobactérias, Staphylococcus aureus, Staphylococcus coagulase negativa, Escherichia coli e Pseudomonas aeruginosa). Já as drogas que apresentaram maiores índices de sensibilidade foram: colistina, polimixina B, amicacina, ciprofloxacino, imipenem, meropenem, linezolida, cefepima, piperacilina+tazobactam, vancomicina e teicoplanina, todas testadas para as mesmas bactérias supracitadas.
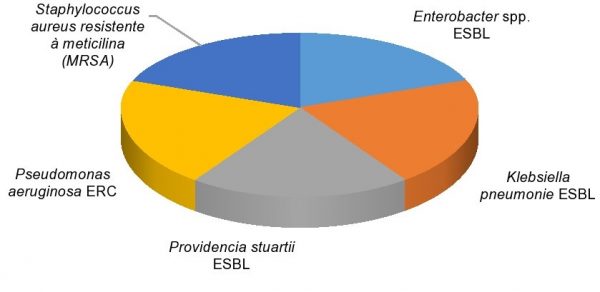
Figura 1. Cepas multirresistentes isoladas na UTI neonatal
Fonte: Dados da pesquisa.
Tabela 3 – Perfil de sensibilidade na média geral para os microrganismos isolados(1)
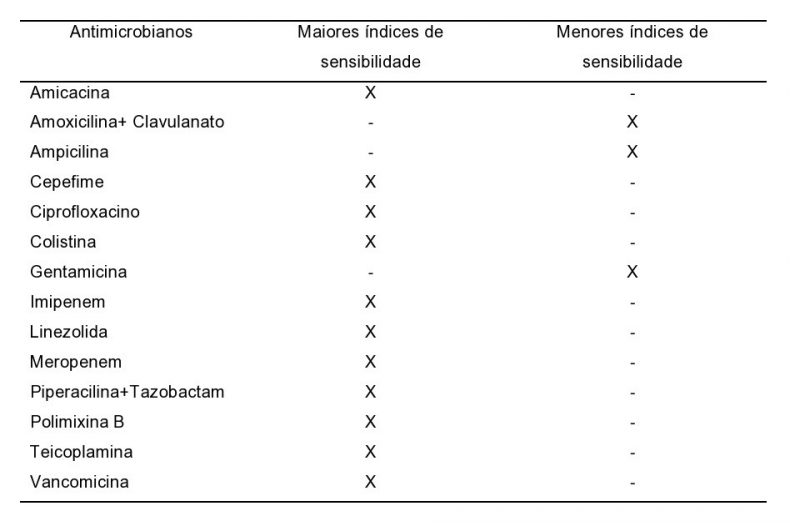
*Esses antimicrobianos foram testados para enterobactérias Staphylococcus aureus, Staphylococcus coagulase negativa, Escherichia coli e Pseudomonas aeruginosa1
Fonte: Dados da pesquisa.
CONCLUSÃO
O uso indiscriminado de antibióticos que vai desde a prescrição até o consumo abusivo desses fármacos pela sociedade é um fator relevante que impulsiona o crescimento das taxas de resistência bacteriana mundialmente. O presente estudo teve como finalidade realizar um levantamento da incidência dos microrganismos isolados em pacientes internos nas Unidades de Terapia Intensiva de um hospital público de grande porte do estado de Sergipe, bem como traçar o perfil de sensibilidade desses patógenos aos antimicrobianos padronizados nesta instituição de saúde. Foi evidenciado através da pesquisa que os números de isolamento de microrganismos tiveram um avanço considerável dentro do período de estudo. Observou-se nesta pesquisa a presença de microrganismos multirresistentes como o Staphylococcus aureus MRSA, Klebsiella pneumoniae ESBL, Enterobacter spp. ESBL, P. aeruginosa ERC e Providencia stuartii ESBL, os quais representam uma preocupação especial para os pacientes imunodeprimidos.
Trabalhos como este são importantes, porque conseguem monitorar os índices de resistência bacteriana no âmbito hospitalar e, por isso, faz-se mister a adoção de medidas educativas voltadas para os diversos profissionais da saúde, como: médicos, farmacêuticos, enfermeiros, bem como à comunidade, no que se refere à conscientização na utilização adequada dos antimicrobianos. É imprescindível que os profissionais de saúde conscientizem-se acerca da sua conduta inadequada no exercício de suas atribuições no cuidado com os pacientes hospitalizados, a qual é responsável pela disseminação de microrganismos, através da contaminação cruzada.
Medidas como a lavagem correta das mãos e a utilização de álcool 70º GL são práticas simples que podem reduzir, consideravelmente, a propagação desses patógenos. A realização de treinamentos, reuniões, palestras e debates para as equipes de saúde e colaboradores na prestação de serviços são alternativas para a divulgação dessas informações, e dessa maneira, contribuir para a redução das taxas de resistência bacteriana. Por fim, é necessário orientar a sociedade sobre os riscos do uso inadequado de antibióticos, como a importância de realizar o tratamento completo, mesmo desaparecendo os sintomas durante os primeiros dias; orientar em relação ao correto descarte desses medicamentos e os prejuízos advindos da automedicação.
REFERÊNCIAS
- Costa ALP, Junior ACSS. Resistência bacteriana aos antibióticos e Saúde Pública: uma breve revisão de literatura. Estação Científica (UNIFAP). 2017; 7(2): p. 45-57.
- Loureiro RJ, Roque F, Rodrigues AT, Herdeiro MT, Ramalheira E. O uso de antibióticos e as resistências bacterianas: breves notas sobre a sua evolucão. Revista Portuguesa Saúde Pública. 2016; 34(1): pp.77-84.
- Pereira SCL, Ribeiro RS, Bazzolli DMS, Vanetti MCD. Resistência a antibióticos e presença de plasmídeos em enterobactérias e Staphylococcus aureus isoladas do setor de dietética de um hospital público. O Mundo da Saúde. 2015; 39(2): p. 147-156
- Santos MJA, Porcy C, Menezes RAO. Etiologia e perfil de resistência bacteriana em uroculturas de pacientes atendidos em um hospital público de Macapá-Amapá, Brasil. Um estudo transversal. Diagnóstico e Tratamento. 2019; 24(4): 135-142.
- Ventola CL. The Antibiotic Resistance Crisis Part 1: Causes and Threats. Pharmacy and Therapeutics. 2015; 40(4): p. 277-283.
- Ribeiro M, Cortina AM. Bactérias de relevância clínica e seus mecanismos de resistência no contexto das infecções relacionadas à assistência a saúde (IRAS). Revista Científica UMC. 2016; 1(1).
- Bordignon JC, Lima LR. Etiologia de infecções hospitalares e perfil de sensibilidade aos antimicrobianos em um hospital do sudoeste do Paraná; Revista Brasileira de Análises Clínicas. 2017; 49(3): p. 283-288.
- Rocha IV, Ferraz PM, Farias TGS, Oliveira SR. Resistência de bactérias isoladas em equipamentos em unidade de terapia intensiva. Associação Caruaruense de Ensino Superior e Técnico. 2015; 28(5): p. 433-439.
- Rodrigues TS, Santos AMR, Lima PC, Moura MEB, Goiano PDOL, Fontinele DRS. Resistência Bacteriana a Antibióticos na Unidade de Terapia Intensiva: Revisão Integrativa. Revista Prevenção de Infecção e Saúde. 2018; 4:7350.
- Santos DVA, Oliveira GA, Pacheco LG, Faria LMO, Cunha JC, Mello TM. Antibióticos através da abordagem do mecanismo de resistência bacteriana. Revista Científica Multidisciplinar das Faculdades São José. 2018; 11(1): p. 02-14.
- Vivas R, Barbosa AAT, Dolabela SS, Jain S. Multidrug-Resistant Bacteria and Alternative Methods to Control Them: An Overview. Microbial Drug Resistance. 2019; 00 (00).
- Oliveira LMS, Santos EM, Costa YGF, Oliveira TL, Rodrigues MML. Perfil de Resistência Bacteriana aos Carbapenêmicos nas Clínicas de um Hospital Universitário. GEP NEWS. 2017; 1(2): p.56-58.
- Modesto EM, Brito DVD. Infecções relacionadas à assistência à saúde em recém-nascidos de alto risco: perfil de resistência dos bacilos Gram negativos. Revista Eletrônica Acervo Saúde. 2019; 11(7).
- Wachholtz MA, Costa AEK, Pissaia LF. Conhecimento dos enfermeiros de uma unidade de terapia intensiva sobre infecções relacionadas à assistência em saúde. Research, Society and Development. 2019; 8(10): e378101397.
- Padoveze MC, Fortaleza CMCB. Infecções associadas à assistência à saúde: desafios para a saúde pública no Brasil. Revista Saúde Pública. 2014; 48(6): p. 995-1001.
- Araújo MQ, Carvalhães RP, Faria SO, Marinho MF, Santos FM, Santos TGQA et al., Perfil de Resistência Bacteriana em Fômites de UTI em Hospital Público do Estado do Tocantins. Revista Cereus. 2017; 9(2): p.126-141.
- Blair JMA, Webber MA, Baylay AJ, Ogbolu DO, Piddock LJV. Molecular mechanisms of antibiotic resistance. Nature Reviews. 2015;13(1): p.42-51.
- Lima CC, Benjamim SCC, Santos RFS. Bacterial resistance mechanism against drugs: a review mecanismo de resistencia bacteriana frente a los fármacos: una revisión. Revista Cuidarte. 2017; 11(1): p. 105-113.
- Cadot L, Bruguière H, Jumas-bilak E, Didelot MN, Masnou A, Barry G. et al., Extended spectrum beta-lactamase-producing Klebsiella pneumoniae outbreak reveals incubators as pathogen reservoir in neonatal care center. European Journal of Pediatrics. 2019; 178(4): p. 505-513.
- Bowen JR, Callander I, Richards R, Lindrea KB. Decreasing infection in neonatal intensive care units through quality improvement. Arch Dis Child Fetal Neonatal. 2017; 102(1): p. 51-57.
- Lima JLC, Alves LR, Paz JNP, Rabelo MA, Macie MAV, Morais MMC. Análise da produção de biofilme por isolados clínicos de Pseudomonas aeruginosa de pacientes com pneumonia associada à ventilação mecânica. Revista Brasileira de Terapia Intensiva. 2017; 29(3): 310-316.
- Leikin-Zach V, Shany E, Yitshak-Sade M, Eshel R, Shafat T, Borer A, et al. Neonatal Risk Factors for Colonization with Extended-Spectrum Beta-Lactamase-Producing Bacteria in the Neonatal Intensive Care Unit. Original Articles. 2018; 5(20): p. 286-290.
- Almeida MVA, Cangussú IM, Carvalho ALS, Brito ILPB, Costa RA. Drug resistance, AmpC-β-lactamase and extended-spectrum β-lactamase-producing Enterobacteriaceae isolated from fish and shrimp. Revista do Instituto de Medicina de São Paulo. 2017; 59.
- Andrade LSA, Carreiro LP, Bastos RPS, Pitanga, TN. Resistência de enterobactérias a Beta-Lactâmicos Mediada por Beta-Lactamases de Espectro Estendido e Carbapenemases. SEMOC.2018.
Correspondência
Henrique Francisco de Almeida
Universidade Tiradentes
Av. Murilo Dantas, 300 – Farolândia, Aracaju – SE,
CEP: 49032-490
E-mail: [email protected]
